SPONGE(Simulation Package tOward Next GEneration molecular modelling)是由北京大学高毅勤课题组开发的分子动力学模拟程序。
¶ 基本介绍
分子动力学(Molecular Dynamics, MD)模拟是化学、物理学、生物学、材料科学和许多其他领域的有用工具。在过去 40 年中,人们开发了各种高效的计算算法和MD程序,用于研究日益复杂和大型系统的动力学,如RNA 聚合酶、细胞膜中的膜蛋白、SARS-CoV-2病毒等。然而,随着应用范围和规模的扩大,分子模拟软件需要更高的计算能力。缩小模拟与实验之间差距的最直接策略是利用更强大的计算硬件。例如,Shaw研究所专门设计了安东(Anton),可以对系统大小为几百万个原子的单结构域蛋白质进行毫秒级模拟。相比之下,使用图形处理单元(GPU)可能是大多数研究小组最经济实惠和最有前途的方法。从另一个方面看,许多先进的计算算法也已开发出来并得到广泛应用,从而延长了模拟时间尺度。特别是在过去几十年中,人们开发了许多增强型采样方法,以实现快速热力学和/或动力学计算。这些方法包括但不限于广泛使用的伞状采样、元动力学、加速MD、复制交换分子动力学(REMD)、并行回火、模拟回火、多正则模拟(特别是通过Wang-Landau算法实现)以及许多其他方法。
在过去的 15 年中,我们致力于开发面向复杂化学和生物系统的高效分子模拟方法,设计了一系列增强型采样方法,实现了构象和轨迹空间的快速采样,并实现了复杂系统热力学和动力学特性的快速计算。 最近,我们开发了一个名为 SPONGE的国产MD模拟软件包,它不仅实现了GPU加速的传统MD模拟,还实现了我们课题组提出的高效增强采样方法。 该软件包具有高度模块化的特点,可以轻松集成其他功能或算法,尤其是最新的深度学习潜力和算法。
¶ 组成部分
目前,SPONGE主要包含三个部分,分别为CudaSPONGE、MindSPONGE和Xponge。
- CudaSPONGE:使用cuda c/c++编写的分子动力学模拟程序
- MindSPONGE:使用神经网络框架Mindspore编写的分子动力学模拟程序
- Xponge:使用python编写的分子动力学模拟前后处理工具
¶ 参考文献
¶ 程序
¶ CudaSPONGE
Yu-Peng Huang, Yijie Xia, Lijiang Yang, Jiachen Wei, Yi Isaac Yang, Yi Qin Gao, SPONGE: A GPU-Accelerated Molecular Dynamics Package with Enhanced Sampling and AI-Driven Algorithms. Chin. J. Chem., 2022, 40: 160-168.
DOI: 10.1002/cjoc.202100456.
¶ MindSPONGE
Jun Zhang, Dechin Chen, Yijie Xia, Yu-Peng Huang, Xiaohan Lin, Xu Han, Ningxi Ni, Zidong Wang, Fan Yu, Lijiang Yang, Yi Isaac Yang, and Yi Qin Gao, Artificial Intelligence Enhanced Molecular Simulations. J. Chem. Theory Comput. 2023 Jul 25;19(14):4338-4350.
DOI: 10.1021/acs.jctc.3c00214.
¶ Xponge
Yijie Xia, and Yi Qin Gao, Xponge: A Python package to perform pre- and post-processing of molecular simulations. J. Open Source Softw., 2022, 7(77), 4467,
DOI: 10.21105/joss.04467
¶ 应用
¶ 2025
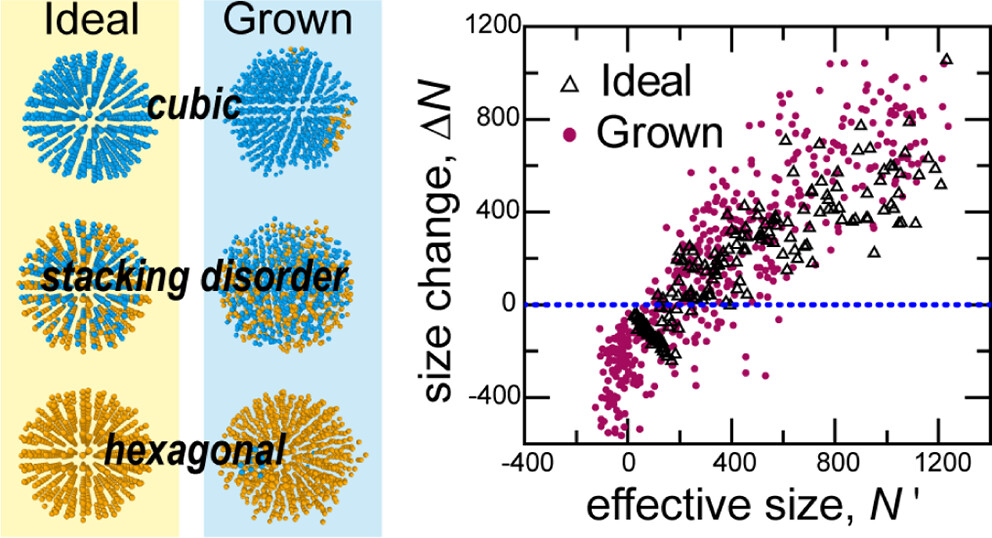
Maodong Li, Yupeng Huang, Yijie Xia, Dechin Chen, Cheng Fan, Lijiang Yang, Yi Qin Gao, and Yi Isaac Yang, Effective Nucleation Size for Ice Crystallization, JCTC, 2025, 21 (4), 1990-1996
DOI: 10.1021/acs.jctc.4c01588
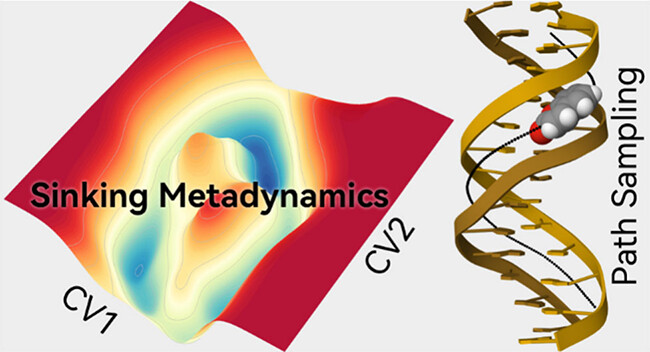
Zhijun Pan, Maodong Li, Dechin Chen, and Yi Isaac Yang, A Sinking Approach to Explore Arbitrary Areas in Free Energy Landscapes,JACS Au Article ASAP,
DOI: 10.1021/jacsau.5c00460
¶ 2024
Qibin Wu, Xinyue Gao, Yifan Lin, Caijin Wu, Jian Zhang, Mengting Chen, Jiaxin Wen, Yajiao Wu, Kun Tian, Wenqiang Bao, Pengming Sun and An Zhu, Integrating Epigenetics, Proteomics, and Metabolomics to Reveal the Involvement of Wnt/β-Catenin Signaling Pathway in Oridonin-Induced Reproductive Toxicity, Toxics, 2024, 12, 339.
DOI: 10.3390/toxics12050339
Yu-Peng Huang, Yijie Xia, Lijiang Yang, and Yi Qin Gao, PMC-IZ: A Simple Algorithm for the Electrostatics Calculation in Slab Geometric Molecular Dynamics Simulations, J. Chem. Theory Comput., 2024, 20, 2, 832–841
DOI: 10.1021/acs.jctc.3c01124
¶ 2023
Ye Tian, Yizhi Song, Yijie Xia, Jiani Hong, Yupeng Huang, Runze Ma, Sifan You, Dong Guan, Duanyun Cao, Mengze Zhao, Ji Chen, Chen Song, Kaihui Liu, Li-Mei Xu, Yi Qin Gao, En-Ge Wang and Ying Jiang, Nanoscale one-dimensional close packing of interfacial alkali ions driven by water-mediated attraction, Nat. Nanotechnol., 2023
DOI: 10.1038/s41565-023-01550-9
Dejia Liu, Hong Zhang, Yu-Peng Huang, and Yi Qin Gao, Investigating the Activation Mechanism Differences between Human and Mouse cGAS by Molecular Dynamics Simulations, J. Phys. Chem. B, 2023, 127, 22, 5034–5045.
DOI: 10.1021/acs.jpcb.3c02377
Shizhi Huang, Yu-Peng Huang, Yijie Xia, Jingyi Ding, Chengyuan Peng, Lulu Wang, Junrong Luo, Xin-Xiang Zhang, Junrong Zheng, Yi Qin Gao, and Jitao Chen, High Li + coordinated solvation sheaths enable high‐quality Li metal anode, InfoMat, 2023, 5(5):e12411.
DOI: 10.1002/inf2.12411
Hongxu Du, Wenjing Zhao, Yijie Xia, Siyu Xie, Yi Tao, Yi Qin Gao, Jie Zhang, and Xinhua Wan, Effect of stereoregularity on excitation-dependent fluorescence and room-temperature phosphorescence of poly(2-vinylpyridine). Aggregate, 2023, 4, e276.
DOI: 10.1002/agt2.276
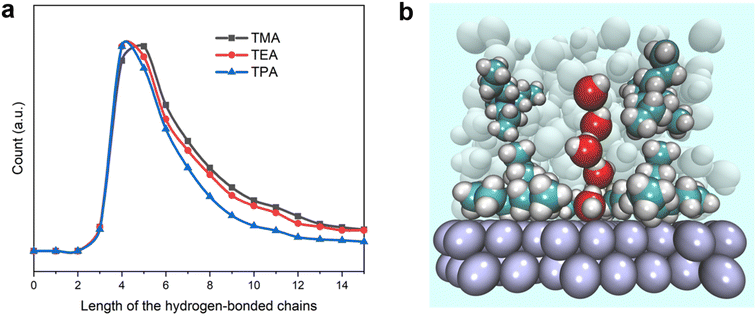
Kaiyue Zhao, Hao Yu, Haocheng Xiong, Qi Lu, Yi Qin Gao and Bingjun Xu, Action at a distance: organic cation induced long range organization of interfacial water enhances hydrogen evolution and oxidation kinetics, Chem. Sci., 2023,14, 11076-11087.
DOI: 10.1039/D3SC03300G
¶ 2022
Wenjing Zhao, Hongxu Du, Yijie Xia, Siyu Xie, Yu-Peng Huang, Tieqi Xu, Jie Zhang, Yi Qin Gao and Xinhua Wan, Accelerating supramolecular aggregation by molecular sliding, Phys. Chem. Chem. Phys., 2022,24, 23840-23848.
DOI: 10.1039/D2CP04064F
¶ 网站更新
¶ 2024
¶ 2024/01/01
推出SPONGE的1.4正式版本
¶ 2023
¶ 2023/12/29
更新网站框架,使用wiki.js
¶ 2023/11/25
更新SPONGE 1.4b0版本
¶ 2023/03/12
更新SPONGE 1.3版本
¶ 2022
¶ 2022/08/13
更新SPONGE 1.2.6版本,支持非周期性边界条件和隐式溶剂的模拟
¶ 2022/04/01
更新SPONGE 1.2.5版本
¶ 2022/01/07
更新SPONGE 1.2beta3版本
¶ 2021
¶ 2021/11/09
更新SPONGE 1.2beta2版本
¶ 2021/10/21
更新SPONGE 1.2beta版本
¶ 2021/08/21
MindSpore SPONGE Workshop成功进行,B站回放
¶ 2021/06/15
网站通过ICP备案和网安备案,上传程序,完成基本搭建